V. Bibliothèques scientifiques avancées▲
V-A. Pandas et xarray▲
P est une bibliothèque pour la structuration et l'analyse avancée de données hétérogènes (PANel DAta). Elle fournit notamment :
- des structures de données relationnelles (« labellisées »), avec une indexation simple ou hiérarchique (c.-à-d. à plusieurs niveaux) ;
- des méthodes d'alignement et d'agrégation des données, avec gestion des données manquantes ;
- un support performant des labels temporels (p.ex. dates, de par son origine dans le domaine de l'économétrie), et des statistiques « glissantes » ;
- de nombreuses fonctions d'entrée/sortie, d'analyse statistiques et de visualisation.
Les fonctionnalités de pandas sont très riches et couvrent de nombreux aspects (données manquantes, dates, analyse statistiques, etc.) : il n'est pas question de toutes les aborder ici. Avant de vous lancer dans une manipulation qui vous semble complexe, bien inspecter la documentation, très complète (p.ex. les recettes du cookbook), pour vérifier qu'elle n'est pas déjà implémentée ou documentée, ou pour identifier l'approche la plus efficace.
La convention utilisée ici est « import pandas as PD ».
Les bibliothèques pandas et xarray sont encore en phase de développement assez intense, et vont probablement être amenées à évoluer significativement, et pas nécessairement de manière rétrocompatible. Nous travaillons ici sur les versions :
V-A-1. Structures▲
Références : Introduction to Data Structures
Pandas dispose de deux grandes structures de données(6) :
|
Nom de la structure |
Rang |
Description |
|---|---|---|
|
1 |
Vecteur de données homogènes labellisées |
|
|
2 |
Tableau structuré de colonnes homogènes |
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
>>> PD.Series(range(3)) # Série d'entiers sans indexation
0 0
1 1
2 2
dtype: int64
>>> PD.Series(N.random.randn(3), index=list('abc')) # Série de réels indexés
a -0.553480
b 0.081297
c -1.845835
dtype: float64
>>> PD.DataFrame(N.random.randn(3, 4))
0 1 2 3
0 1.570977 -0.677845 0.094364 -0.362031
1 -0.136712 0.762300 0.068611 1.265816
2 -0.697760 0.791288 0.449645 -1.105062
>>> PD.DataFrame([(1, N.exp(1), 'un'), (2, N.exp(2), 'deux'), (3, N.exp(3), 'trois')],
... index=list('abc'), columns='i val nom'.split())
i val nom
a 1 2.718282 un
b 2 7.389056 deux
c 3 20.085537 trois
Pour mettre en évidence la puissance de Pandas, nous utiliserons le catalogue des objets Messier vu précédemment. Le fichier peut être importé à l'aide de la fonction pandas.read_csv(), et le dataframe résultant est labellisé à la volée par la colonne M (pandas.DataFrame.set_index()) :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
>>> messier = PD.read_csv("Messier.csv", comment='#') # Lecture du fichier CSV
>>> messier.set_index('M', inplace=True) # Indexation sur la colonne "M"
>>> messier.info() # Informations générales
<class 'pandas.core.frame.DataFrame'>
Index: 110 entries, M1 to M110
Data columns (total 10 columns):
NGC 108 non-null object
Type 110 non-null object
Mag 110 non-null float64
Size 110 non-null float64
Distance 110 non-null float64
RA 110 non-null float64
Dec 110 non-null float64
Con 110 non-null object
Season 110 non-null object
Name 31 non-null object
dtypes: float64(5), object(5)
memory usage: 9.5+ KB
>>> messier.head(3) # Par défaut les 5 premières lignes
NGC Type Mag Size Distance RA Dec Con Season Name
M
M1 1952 Sn 8.4 5.0 1930.0 5.575 22.017 Tau winter Crab Nebula
M2 7089 Gc 6.5 12.9 11600.0 21.558 0.817 Aqr autumn NaN
M3 5272 Gc 6.2 16.2 10400.0 13.703 28.383 CVn spring NaN
Un dataframe est caractérisé par son indexation pandas.DataFrame.index et ses colonnes pandas.DataFrame.columns (de type pandas.Index ou pandas.MultiIndex), et les valeurs des données pandas.DataFrame.values :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
25.
26.
>>> messier.index # Retourne un Index
Index([u'M1', u'M2', u'M3', ..., u'M108', u'M109', u'M110'],
dtype='object', name=u'M', length=110)
>>> messier.columns # Retourne un Index
Index([u'NGC', u'Type', u'Mag', ..., u'Con', u'Season', u'Name'],
dtype='object')
>>> messier.dtypes # Retourne une Series indexée sur le nom des colonnes
NGC object
Type object
Mag float64
Size float64
Distance float64
RA float64
Dec float64
Con object
Season object
Name object
dtype: object
>>> messier.values
array([['1952', 'Sn', 8.4, ..., 'Tau', 'winter', 'Crab Nebula'],
['7089', 'Gc', 6.5, ..., 'Aqr', 'autumn', nan],
...,
['3992', 'Ba', 9.8, ..., 'UMa', 'spring', nan],
['205', 'El', 8.5, ..., 'And', 'autumn', nan]], dtype=object)
>>> messier.shape
(110, 10)
Une description statistique sommaire des colonnes numériques est obtenue par pandas.DataFrame.describe() :
2.
3.
4.
5.
6.
7.
8.
9.
10.
>>> messier.drop(['RA', 'Dec'], axis=1).describe()
Mag Size Distance
count 110.000000 110.000000 1.100000e+02
mean 7.492727 17.719091 4.574883e+06
std 1.755657 22.055100 7.141036e+06
min 1.600000 0.800000 1.170000e+02
25% 6.300000 6.425000 1.312500e+03
50% 7.650000 9.900000 8.390000e+03
75% 8.900000 17.300000 1.070000e+07
max 10.200000 120.000000 1.840000e+07
V-A-2. Accès aux données▲
Référence : Indexing and Selecting Data
L'accès par colonne retourne une pandas.Series (avec la même indexation) pour une colonne unique, ou un nouveau pandas.DataFrame pour plusieurs colonnes :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
>>> messier.NGC # Équivalent à messier['NGC']
M
M1 1952
M2 7089
...
M109 3992
M110 205
Name: NGC, Length: 110, dtype: object
>>> messier[['RA', 'Dec']] # = messier.filter(items=('RA', 'Dec'))
RA Dec
M
M1 5.575 22.017
M2 21.558 0.817
... ... ...
M109 11.960 53.383
M110 0.673 41.683
[110 rows x 2 columns]
L'accès par slice retourne un nouveau dataframe :
2.
3.
4.
5.
6.
>>> messier[:6:2] # Lignes 0 (inclus) à 6 (exclu) par pas de 2
NGC Type Mag Size Distance RA Dec Con Season Name
M
M1 1952 Sn 8.4 5.0 1930.0 5.575 22.017 Tau winter Crab Nebula
M3 5272 Gc 6.2 16.2 10400.0 13.703 28.383 CVn spring NaN
M5 5904 Gc 5.6 17.4 7520.0 15.310 2.083 Ser summer NaN
L'accès peut également se faire par labels via pandas.DataFrame.loc :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
>>> messier.loc['M31'] # Retourne une Series indexée par les noms de colonne
NGC 224
Type Sp
...
Season autumn
Name Andromeda Galaxy
Name: M31, Length: 10, dtype: object
>>> messier.loc['M31', ['Type', 'Name']] # Retourne une Series
Type Sp
Name Andromeda Galaxy
Name: M31, dtype: object
>>> messier.loc[['M31', 'M51'], ['Type', 'Name']] # Retourne un DataFrame
Type Name
M
M31 Sp Andromeda Galaxy
M51 Sp Whirlpool Galaxy
>>> messier.loc['M31':'M33', ['Type', 'Name']] # De M31 à M33 inclu
Type Name
M
M31 Sp Andromeda Galaxy
M32 El NaN
M33 Sp Triangulum Galaxy
De façon symétrique, l'accès peut se faire par position (n° de ligne/colonne) via pandas.DataFrame.iloc, p.ex. :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
>>> messier.iloc[30:33, [1, -1]] # Ici, l'indice 33 n'est PAS inclus !
Type Name
M
M31 Sp Andromeda Galaxy
M32 El NaN
M33 Sp Triangulum Galaxy
>>> messier.iloc[30:33, messier.columns.get_loc('Name')] # Mix des 2 approches
M
M31 Andromeda Galaxy
M32 NaN
M33 Triangulum Galaxy
Name: Name, dtype: object
Les fonctions pandas.DataFrame.at et pandas.DataFrame.iat permettent d'accéder rapidement aux données individuelles :
2.
3.
4.
>>> messier.at['M31', 'NGC'] # 20× plus rapide que messier.loc['M31']['NGC']
'224'
>>> messier.iat[30, 0] # 20× plus rapide que messier.iloc[0][0]
'224'
Noter qu'il existe une façon de filtrer les données sur les colonnes ou les labels :
Comme pour numpy, il est possible d'opérer une sélection booléenne :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
25.
26.
27.
28.
29.
30.
31.
32.
33.
34.
35.
>>> messier.loc[messier['Con'] == 'UMa', ['NGC', 'Name']]
NGC Name
M
M40 Win4 Winnecke 4
M81 3031 Bode's Galaxy
M82 3034 Cigar Galaxy
M97 3587 Owl Nebula
M101 5457 Pinwheel Galaxy
M108 3556 NaN
M109 3992 NaN
>>> messier[messier['Season'].isin('winter spring'.split())].head(3)
NGC Type Mag Size Distance RA Dec Con Season Name
M
M1 1952 Sn 8.4 5.0 1930.0 5.575 22.017 Tau winter Crab Nebula
M3 5272 Gc 6.2 16.2 10400.0 13.703 28.383 CVn spring NaN
M35 2168 Oc 5.3 28.0 859.0 6.148 24.333 Gem winter NaN
>>> messier.loc[lambda df: N.radians(df.Size / 60) * df.Distance < 1].Name
M
M27 Dumbbell Nebula
M40 Winnecke 4
M57 Ring Nebula
M73 NaN
M76 Little Dumbbell Nebula
M78 NaN
M97 Owl Nebula
Name: Name, dtype: object
>>> messier.query("(Mag < 5) & (Size > 60)").Name
M
M7 Ptolemy's Cluster
M24 Sagittarius Star Cloud
M31 Andromeda Galaxy
M42 Great Nebula in Orion
M44 Beehive Cluster
M45 Pleiades
Name: Name, dtype: object
|
Sélection |
Syntaxe |
Résultat |
|---|---|---|
|
Colonne unique |
df.col |
|
|
Liste de colonnes |
df[[c1, ...]] |
|
|
Lignes par tranche |
df[slice] |
|
|
Label unique |
df.loc[label] |
|
|
Liste de labels |
df.loc[[lab1, ...]] |
|
|
Labels par tranche |
df.loc[lab1:lab2] |
|
|
Ligne entière par n° |
df.iloc[i] |
|
|
Ligne partielle par n° |
df.iloc[i, [j,...]] |
|
|
Valeur par label |
df.at[lab, col] |
Scalaire |
|
Valeur par n° |
df.iat[i, j] |
Scalaire |
|
Ligne par sél. booléenne |
df.loc[sel] |
pandas.DataFrame.drop() permet d'éliminer une ou plusieurs colonnes d'un dataframe :
2.
3.
4.
5.
6.
>>> messier.drop(['RA', 'Dec'], axis=1).head(3) # Élimination de colonnes
NGC Type Mag Size Distance Con Season Name
M
M1 1952 Sn 8.4 5.0 1930.0 Tau winter Crab Nebula
M2 7089 Gc 6.5 12.9 11600.0 Aqr autumn NaN
M3 5272 Gc 6.2 16.2 10400.0 CVn spring NaN
pandas.DataFrame.dropna() et pandas.DataFrame.fillna() permettent de gérer les données manquantes (NaN) :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
>>> messier.dropna(axis=0, how='any', subset=['NGC', 'Name']).head(3)
NGC Type Mag Size Distance RA Dec Con Season Name
M
M1 1952 Sn 8.4 5.0 1930.0 5.575 22.017 Tau winter Crab Nebula
M6 6405 Oc 4.2 25.0 491.0 17.668 -32.217 Sco summer Butterfly Cluster
M7 6475 Oc 3.3 80.0 245.0 17.898 -34.817 Sco summer Ptolemy's Cluster
>>> messier.fillna('', inplace=True) # Remplace les NaN à la volée
>>> messier.head(3)
NGC Type Mag Size Distance RA Dec Con Season Name
M
M1 1952 Sn 8.4 5.0 1930.0 5.575 22.017 Tau winter Crab Nebula
M2 7089 Gc 6.5 12.9 11600.0 21.558 0.817 Aqr autumn
M3 5272 Gc 6.2 16.2 10400.0 13.703 28.383 CVn spring
Référence : Working with missing data
Attention par défaut, beaucoup d'opérations retournent une copie de la structure, sauf si l'opération se fait « sur place » (inplace=True). D'autres opérations d'accès retournent seulement une nouvelle vue des mêmes données.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
>>> df = PD.DataFrame(N.arange(12).reshape(3, 4),
... index=list('abc'), columns=list('ABCD')); df
A B C D
a 0 1 2 3
b 4 5 6 7
c 8 9 10 11
>>> df.drop('a', axis=0)
A B C D
b 4 5 6 7
c 8 9 10 11
>>> colA = df['A'] # Nouvelle vue de la colonne 'A'
>>> colA += 1 # Opération sur place
>>> df # la ligne 'a' est tjs là, la colonne 'A' a été modifiée
A B C D
a 1 1 2 3
b 5 5 6 7
c 9 9 10 11
Indexation hiérarchique
Références : Multiindex / Advanced Indexing
pandas.MultiIndex offre une indexation hiérarchique, permettant de stocker et manipuler des données avec un nombre arbitraire de dimensions dans des structures plus simples.
2.
3.
4.
5.
6.
7.
8.
>>> saisons = messier.reset_index() # Élimine l'indexation actuelle
>>> saisons.set_index(['Season', 'Type'], inplace=True) # MultiIndex
>>> saisons.head(3)
M NGC Mag Size Distance RA Dec Con Name
Season Type
winter Sn M1 1952 8.4 5.0 1930.0 5.575 22.017 Tau Crab Nebula
autumn Gc M2 7089 6.5 12.9 11600.0 21.558 0.817 Aqr
spring Gc M3 5272 6.2 16.2 10400.0 13.703 28.383 CVn
Les informations contenues sont toujours les mêmes, mais structurées différemment :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
>>> saisons.loc['spring'].head(3) # Utilisation du 1er label
M NGC Mag Size Distance RA Dec Con Name
Type
Gc M3 5272 6.2 16.2 10400.0 13.703 28.383 CVn
Ds M40 Win4 8.4 0.8 156.0 12.373 58.083 UMa Winnecke 4
El M49 4472 8.4 8.2 18400000.0 12.497 8.000 Vir
>>> saisons.loc['spring', 'El'].head(3) # Utilisation des 2 labels
M NGC Mag Size Distance RA Dec Con Name
Season Type
spring El M49 4472 8.4 8.2 18400000.0 12.497 8.00 Vir
El M59 4621 9.6 4.2 18400000.0 12.700 11.65 Vir
El M60 4649 8.8 6.5 18400000.0 12.728 11.55 Vir
La fonction pandas.DataFrame.xs() permet des sélections sur les différents niveaux d'indexation :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
>>> saisons.xs('spring', level='Season').head(3) # = saisons.loc['spring']
M NGC Mag Size Distance RA Dec Con Name
Type
Gc M3 5272 6.2 16.2 10400.0 13.703 28.383 CVn
Ds M40 Win4 8.4 0.8 156.0 12.373 58.083 UMa Winnecke 4
El M49 4472 8.4 8.2 18400000.0 12.497 8.000 Vir
>>> saisons.xs('El', level='Type').head(3) # Sélection sur le 2e niveau
M NGC Mag Size Distance RA Dec Con Name
Season
autumn M32 221 8.1 7.0 920000.0 0.713 40.867 And
spring M49 4472 8.4 8.2 18400000.0 12.497 8.000 Vir
spring M59 4621 9.6 4.2 18400000.0 12.700 11.650 Vir
Le (multi)index n'est pas nécessairement trié à sa création, pandas.sort_index() permet d'y remédier :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
>>> saisons[['M', 'NGC', 'Name']].head()
M NGC Name
Season Type
winter Sn M1 1952 Crab Nebula
autumn Gc M2 7089
spring Gc M3 5272
summer Gc M4 6121
Gc M5 5904
>>> saisons[['M', 'NGC', 'Name']].sort_index().head()
M NGC Name
Season Type
autumn El M32 221
El M110 205
Gc M2 7089
Gc M15 7078 Great Pegasus Globular
Gc M30 7099
V-A-3. Manipulation des données▲
Références : Essential Basic Functionality
Comme dans numpy, il est possible de modifier les valeurs, ajouter/retirer des colonnes ou des lignes, tout en gérant les données manquantes.
l'interopérabilité entre pandas et numpy est totale, toutes les fonctions Numpy peuvent prendre une structure Pandas en entrée, et s'appliquer aux colonnes appropriées :
2.
3.
4.
5.
6.
7.
>>> N.mean(messier, axis=0) # Moyenne sur les lignes ? Series indexée sur les colonnes
Mag 7.492727e+00
Size 1.771909e+01
Distance 4.574883e+06
RA 1.303774e+01
Dec 9.281782e+00
dtype: float64
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
25.
26.
27.
28.
29.
>>> N.random.seed(0)
>>> df = PD.DataFrame(
{'one': PD.Series(N.random.randn(3), index=['a', 'b', 'c']),
'two': PD.Series(N.random.randn(4), index=['a', 'b', 'c', 'd']),
'three': PD.Series(N.random.randn(3), index=['b', 'c', 'd'])})
>>> df
one three two
a 1.764052 NaN 2.240893
b 0.400157 -0.151357 1.867558
c 0.978738 -0.103219 -0.977278
d NaN 0.410599 0.950088
>>> df['four'] = df['one'] + df['two']; df # Création d'une nouvelle colonne
one three two four
a 1.764052 NaN 2.240893 4.004946
b 0.400157 -0.151357 1.867558 2.267715
c 0.978738 -0.103219 -0.977278 0.001460
d NaN 0.410599 0.950088 NaN
>>> df.sub(df.loc['b'], axis='columns') # Soustraction d'une ligne à toutes les colonnes (axis=1)
one three two four
a 1.363895 NaN 0.373335 1.737230
b 0.000000 0.000000 0.000000 0.000000
c 0.578581 0.048138 -2.844836 -2.266255
d NaN 0.561956 -0.917470 NaN
>>> df.sub(df['one'], axis='index') # Soustraction d'une colonne à toutes les lignes (axis=0 ou 'rows')
one three two four
a 0.0 NaN 0.476841 2.240893
b 0.0 -0.551514 1.467401 1.867558
c 0.0 -1.081957 -1.956016 -0.977278
d NaN NaN NaN NaN
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
>>> df.sort_values(by='a', axis=1) # Tri des colonnes selon les valeurs de la ligne 'a'
one two three
a 1.764052 2.240893 NaN
b 0.400157 1.867558 -0.151357
c 0.978738 -0.977278 -0.103219
d NaN 0.950088 0.410599
>>> df.min(axis=1) # Valeur minimale le long des colonnes
a 1.764052
b -0.151357
c -0.977278
d 0.410599
dtype: float64
>>> df.idxmin(axis=1) # Colonne des valeurs minimales le long des colonnes
a one
b three
c two
d three
dtype: object
2.
3.
4.
5.
>>> df.mean(axis=0) # Moyenne sur toutes les lignes (gestion des données manquantes)
one 1.047649
three 0.052007
two 1.020315
dtype: float64
Si les bibliothèques d'optimisation de performances Bottleneck et NumExpr sont installées, pandas en bénéficiera de façon transparente.
V-A-4. Regroupement et agrégation de données▲
Histogramme et discrétisation
Compter les objets Messier par constellation avec pandas.value_counts() :
2.
3.
4.
5.
>>> PD.value_counts(messier['Con']).head(3)
Sgr 15
Vir 11
Com 8
Name: Con, dtype: int64
Partitionner les objets en trois groupes de magnitude (par valeurs : pandas.cut(), par quantiles : pandas.qcut()), et les compter :
2.
3.
4.
5.
6.
7.
8.
9.
10.
>>> PD.value_counts(PD.cut(messier['Mag'], 3)).sort_index() # Par valeurs
(1.591, 4.467] 6
(4.467, 7.333] 40
(7.333, 10.2] 64
Name: Mag, dtype: int64
>>> PD.value_counts(PD.qcut(messier['Mag'], [0, .3, .6, 1])).sort_index() # Par quantiles
(1.599, 6.697] 36
(6.697, 8.4] 38
(8.4, 10.2] 36
Name: Mag, dtype: int64
Group-by
Référence : Group By: split-apply-combine
Pandas offre la possibilité de regrouper les données selon divers critères (pandas.DataFrame.groupby()), de les agréger au sein de ces groupes et de stocker les résultats dans une structure avec indexation hiérarchique (pandas.DataFrame.agg()).
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
25.
26.
27.
28.
29.
30.
31.
32.
33.
34.
35.
36.
37.
>>> seasonGr = messier.groupby('Season') # Retourne un DataFrameGroupBy
>>> seasonGr.groups
{'autumn': Index([u'M2', u'M15', ..., u'M103', u'M110'],
dtype='object', name=u'M'),
'spring': Index([u'M3', u'M40', ..., u'M108', u'M109'],
dtype='object', name=u'M'),
'summer': Index([u'M4', u'M5', ..., u'M102', u'M107'],
dtype='object', name=u'M'),
'winter': Index([u'M1', u'M35', ..., u'M79', u'M93'],
dtype='object', name=u'M')}
>>> seasonGr.size()
Season
autumn 14
spring 38
summer 40
winter 18
dtype: int64
>>> seasonGr.get_group('winter').head(3)
Con Dec Distance Mag NGC Name RA Size Type
M
M1 Tau 22.017 1930.0 8.4 1952 Crab Nebula 5.575 5.0 Sn
M35 Gem 24.333 859.0 5.3 2168 6.148 28.0 Oc
M36 Aur 34.133 1260.0 6.3 1960 5.602 12.0 Oc
>>> seasonGr['Size'].agg([N.mean, N.std]) # Taille moyenne et stddev par groupe
mean std
Season
autumn 24.307143 31.472588
spring 7.197368 4.183848
summer 17.965000 19.322400
winter 34.261111 29.894779
>>> seasonGr.agg({'Size': N.max, 'Mag': N.min})
Mag Size
Season
autumn 3.4 120.0
spring 6.2 22.0
summer 3.3 90.0
winter 1.6 110.0
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
>>> magGr = messier.groupby(
... [PD.qcut(messier['Mag'], [0, .3, .6, 1],
... labels='Bright Medium Faint'.split()),
... "Season"])
>>> magGr['Mag', 'Size'].agg({'Mag': ['count', 'mean'],
... 'Size': [N.mean, N.std]})
Mag Size
count mean mean std
Mag Season
Bright autumn 6 5.316667 45.200000 40.470878
spring 1 6.200000 16.200000 NaN
summer 15 5.673333 30.840000 26.225228
winter 13 5.138462 42.923077 30.944740
Faint autumn 4 9.225000 8.025000 4.768910
spring 30 9.236667 5.756667 2.272578
summer 7 8.971429 7.814286 9.135540
winter 3 8.566667 9.666667 6.429101
Medium autumn 4 7.500000 9.250000 3.304038
spring 7 7.714286 12.085714 5.587316
summer 18 7.366667 11.183333 4.825453
winter 2 7.550000 14.850000 8.697413
Tableau croisé (Pivot table)
Référence : Reshaping and Pivot Tables
Calculer la magnitude et la taille moyennes des objets Messier selon leur type avec pandas.DataFrame.pivot_table() :
2.
3.
4.
>>> messier['Mag Size Type'.split()].pivot_table(columns='Type')
Type As Ba Di ... Pl Sn Sp
Mag 9.0 9.85 7.216667 ... 9.050 8.4 8.495652
Size 2.8 4.80 33.333333 ... 3.425 5.0 15.160870
V-A-5. Visualisations▲
Exemple :
Démonstration Pandas/Seaborn (notebook : pokemon.ipynb) sur le jeu de données : Pokemon.csv.
Références :
Autres exemples de visualisation de jeux de données complexes (utilisation de pandas et seaborn)
Liens
- Pandas Tutorial
- Pandas Cookbook
- Pandas Lessons for New Users
- Practical Data Analysis
- Modern Pandas
- Various tutorials
Exercices :
V-A-6. Xarray▲
xarray est une bibliothèque pour la structuration de données homogènes de dimension arbitraire. Suivant la philosophie de la bibliothèque Pandas dont elle est issue (et dont elle dépend), elle permet notamment de nommer les différentes dimensions (axes) des tableaux (p.ex. x.sum(axis='time')), d'indexer les données (p.ex. x.loc['M31']), de naturellement gérer les opérations de broadcasting, des opérations de regroupement et d'agrégation (p.ex. x.groupby(time.dayofyear).mean()), une gestion plus facile des données manquantes et d'alignement des tableaux indexés (p.ex. align(x, y, join='outer')).
pandas excels at working with tabular data. That suffices for many statistical analyses, but physical scientists rely on N-dimensional arrays - which is where xarray comes in.
xarray fournit deux structures principales, héritées du format netCDF :
- xarray.DataArray, un tableau N-D indexé généralisant le pandas.Series ;
- xarray.DataSet, un dictionnaire regroupant plusieurs DataArray alignés selon une ou plusieurs dimensions, et similaire au pandas.DataFrame.
La convention utilisée ici est « import xarray as X ».
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
25.
26.
27.
28.
29.
30.
31.
32.
33.
34.
35.
36.
37.
38.
39.
40.
41.
42.
43.
44.
45.
46.
47.
48.
>>> N.random.seed(0)
>>> data = X.DataArray(N.arange(3*4, dtype=float).reshape((3, 4)), # Tableau de données
... dims=('x', 'y'), # Nom des dimensions
... coords={'x': list('abc')}, # Indexation des coordonnées en 'x'
... name='mesures', # Nom du tableau
... attrs=dict(author='Y. Copin')) # Métadonnées
>>> data
<xarray.DataArray 'mesures' (x: 3, y: 4)>
array([[ 0., 1., 2., 3.],
[ 4., 5., 6., 7.],
[ 8., 9., 10., 11.]])
Coordinates:
* x (x) |S1 'a' 'b' 'c'
Dimensions without coordinates: y
Attributes:
author: Y. Copin
>>> data.to_pandas() # Conversion en DataFrame à indexation simple
y 0 1 2 3
x
a 0.0 1.0 2.0 3.0
b 4.0 5.0 6.0 7.0
c 8.0 9.0 10.0 11.0
>>> data.to_dataframe() # Conversion en DataFrame multi-indexé (hiérarchique)
mesures
x y
a 0 0.0
1 1.0
2 2.0
3 3.0
b 0 4.0
1 5.0
2 6.0
3 7.0
c 0 8.0
1 9.0
2 10.0
3 11.0
>>> data.dims
('x', 'y')
>>> data.coords
Coordinates:
* x (x) |S1 'a' 'b' 'c'
>>> data.values
array([[ 0., 1., 2., 3.],
[ 4., 5., 6., 7.],
[ 8., 9., 10., 11.]])
>>> data.attrs
OrderedDict([('author', 'Y. Copin')])
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
>>> data[:, 1] # Accès par indices
<xarray.DataArray 'mesures' (x: 3)>
array([ 1., 5., 9.])
Coordinates:
* x (x) |S1 'a' 'b' 'c'
>>> data.loc['a':'b', -1] # Accès par labels
<xarray.DataArray 'mesures' (x: 2)>
array([ 3., 7.])
Coordinates:
* x (x) |S1 'a' 'b'
>>> data.sel(x=['a', 'c'], y=2)
<xarray.DataArray 'mesures' (x: 2)>
array([ 2., 10.])
Coordinates:
* x (x) |S1 'a' 'c'
2.
3.
4.
>>> data.mean(dim='x') # Moyenne le long de la dimension 'x' = data.mean(axis=0)
<xarray.DataArray 'mesures' (y: 4)>
array([ 4., 5., 6., 7.])
Dimensions without coordinates: y
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
>>> data2 = X.DataArray(N.arange(6).reshape(2, 3) * 10,
... dims=('z', 'x'), coords={'x': list('abd')})
>>> data2.to_pandas()
x a b d
z
0 0 10 20
1 30 40 50
>>> data.to_pandas() # REMINDER
y 0 1 2 3
x
a 0.0 1.0 2.0 3.0
b 4.0 5.0 6.0 7.0
c 8.0 9.0 10.0 11.0
>>> data2.values + data.values # Opération sur des tableaux numpy incompatibles
ValueError: operands could not be broadcast together with shapes (2,3) (3,4)
>>> data2 + data # Alignement automatique sur les coord. communes !
<xarray.DataArray (z: 2, x: 2, y: 4)>
array([[[ 0., 1., 2., 3.],
[ 14., 15., 16., 17.]],
[[ 30., 31., 32., 33.],
[ 44., 45., 46., 47.]]])
Coordinates:
* x (x) object 'a' 'b'
Dimensions without coordinates: z, y
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
>>> data['isSmall'] = data.sum(dim='y') < 10; data # Booléen "Somme sur y < 10"
<xarray.DataArray 'mesures' (x: 3, y: 4)>
array([[ 0., 1., 2., 3.],
[ 4., 5., 6., 7.],
[ 8., 9., 10., 11.]])
Coordinates:
* x (x) |S1 'a' 'b' 'c'
isSmall (x) bool True False False
Dimensions without coordinates: y
>>> data.groupby('isSmall').mean(dim='x') # Regroupement et agrégation
<xarray.DataArray 'mesures' (isSmall: 2, y: 4)>
array([[ 6., 7., 8., 9.],
[ 0., 1., 2., 3.]])
Coordinates:
* isSmall (isSmall) object False True
Dimensions without coordinates: y
Exemples plus complexes :
N'oubliez pas de citer xarray dans vos publications et présentations.
V-B. Astropy▲
Astropy est une bibliothèque astronomique maintenue par la communauté et visant à fédérer les efforts jusque-là disparates. Elle offre en outre une interface unifiée à des bibliothèques affiliées plus spécifiques.
V-B-1. Tour d'horizon▲
-
Structures de base :
- astropy.constants : constantes fondamentales (voir également scipy.constants) ;
- astropy.units : unités et quantités dimensionnées ;
- astropy.nddata : extension des numpy.ndarray (incluant métadonnées, masque, unité, incertitude, etc.) ;
- astropy.table : tableaux hétérogènes ;
- astropy.time : manipulation du temps et des dates ;
- astropy.coordinates : systèmes de coordonnées ;
- astropy.wcs : World Coordinate System ;
- astropy.modeling : modèles et ajustements ;
- astropy.analytic_functions : fonctions analytiques.
-
Entrées/sorties :
- astropy.io.fits : fichiers FITS ;
- astropy.io.ascii : tables ASCII ;
- astropy.io.votable : XML VO-tables ;
- astropy.io.misc : divers ;
- astropy.vo : accès au Virtual Observatory.
-
Calculs astronomiques :
- astropy.cosmology : calculs cosmologiques ;
- astropy.convolution : convolution et filtrage ;
- astropy.visualization : visualisation de données ;
- astropy.stats : méthodes astrostatistiques.
V-B-2. Démonstration▲
Démonstration AstropyDémonstration (astropy.ipynb)
Nous présentons ici quelques possibilités de la bibliothèque Astropy.
Référence : cette démonstration est très largement inspirée de la partie Astropy du cours Python Euclid 2016.
In [1] :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
# Mimic Python-3.x
from __future__ import print_function, division
import numpy as N
import matplotlib.pyplot as P
try:
import seaborn
seaborn.set_color_codes() # Override default matplotlib colors 'b', 'r', 'g', etc.
except ImportError:
pass
# Interactive figures
# %matplotlib notebook
# Static figures
%matplotlib inline
V-B-2-a. Fichiers FITS▲
Le format FITS (Flexible Image Transport System) constitue le format de données historique (et encore très utilisé) de la communauté astronomique. Il permet le stockage simultané de données - sous forme de tableaux numériques multidimensionnels (spectre 1D, image 2D, cube 3D, etc.) ou de tables de données structurées (texte ou binaires) - et des métadonnées associées - sous la forme d'un entête ASCII nommé header. Il autorise en outre de combiner au sein d'un même fichier différents segments de données (extensions, p.ex. le signal et la variance associée) sous la forme de HDU (Header-Data Units).
Le fichier FITS de test est disponible ici : image.fits (données Herschel Space Observatory).
V-B-2-a-i. Lire un fichier FITS▲
In [2] :
2.
3.
4.
from astropy.io import fits as F
filename = "image.fits"
hdulist = F.open(filename)
Hdulist est un objet HDUList de type liste regroupant les différents HDU du fichier :
In [3] :
hdulist.info()
Filename: image.fits
No. Name Type Cards Dimensions Format
0 PRIMARY PrimaryHDU 151 ()
1 image ImageHDU 52 (273, 296) float64
2 error ImageHDU 20 (273, 296) float64
3 coverage ImageHDU 20 (273, 296) float64
4 History ImageHDU 23 ()
5 HistoryScript BinTableHDU 39 105R x 1C [300A]
6 HistoryTasks BinTableHDU 46 77R x 4C [1K, 27A, 1K, 9A]
7 HistoryParameters BinTableHDU 74 614R x 10C [1K, 20A, 7A, 46A, 1L, 1K, 1L, 74A, 11A, 41A]Chaque HDU contient:
- un attribut data pour la partie données sous la forme d'un numpy.array ou d'une structure équivalente à un tableau à type structuré ;
- un attribut header pour la partie métadonnées sous la forme “ KEY
=value/comment ”.
In [4] :
Out [4] :
<type 'numpy.ndarray'> <class 'astropy.io.fits.header.Header'>
Il est également possible de lire directement les données et les métadonnées de l'extension image :
In [5] :
Out [5] :
<type 'numpy.ndarray'> <class 'astropy.io.fits.header.Header'>
data contient donc les données numériques, ici un tableau 2D :
In [6] : N.info(ima)
Out [6] :
class: ndarray
shape: (296, 273)
strides: (2184, 8)
itemsize: 8
aligned: True
contiguous: True
fortran: False
data pointer: 0x7f43bce3bec0
byteorder: big
byteswap: True
type: >f8L'entête hdr est un objet de type Header similaire à un OrderedDict (dictionnaire ordonné).
In [7] : hdr[:5] # Les 5 premières clés de l'entête
Out [7] :
XTENSION= 'IMAGE ' / Java FITS: Wed Aug 14 11:37:21 CEST 2013
BITPIX = -64
NAXIS = 2 / Dimensionality
NAXIS1 = 273
NAXIS2 = 296Les axes des tableaux FITS et NumPy arrays sont inversés !
In [8] :
print("FITS: ", (hdr['naxis1'], hdr['naxis2'])) # format de l'image FITS
print("Numpy:", ima.shape) # format du tableau numpyOut [8] :
FITS: (273, 296)
Numpy: (296, 273)V-B-2-a-ii. World Coordinate System▲
L'entête d'un fichier FITS peut notamment inclure une description détaillée du système de coordonnées lié aux données, le World Coordinate System.
In [9] :
2.
3.
4.
from astropy import wcs as WCS
wcs = WCS.WCS(hdr) # Décrypte le WCS à partir de l'entête
print(wcs)
Out [9] :
WCS Keywords
Number of WCS axes: 2
CTYPE : 'RA---TAN' 'DEC--TAN'
CRVAL : 30.07379502155236 -24.903630299920962
CRPIX : 134.0 153.0
NAXIS : 273 296In [10] :
2.
3.
4.
ra, dec = wcs.wcs_pix2world(0, 0, 0) # Coordonnées réelles du px (0, 0)
print("World:", ra, dec)
x, y = wcs.wcs_world2pix(ra, dec, 0) # Coordonnées dans l'image de la position (ra, dec)
print("Image:", x, y)
Out [10] :
World: 30.318687003 -25.1567606072
Image: -3.21165316564e-12 -1.05160324892e-12V-B-2-a-iii. Visualisation dans matplotlib▲
Les tableaux 2D (image) se visualisent à l'aide de la commande imshow :
- cmap : table des couleurs ;
- vmin, vmax : valeurs minimale et maximale des données visualisées ;
- origin : position du pixel (0, 0) (‘lower' = en bas à gauche).
In [11] :
2.
3.
4.
5.
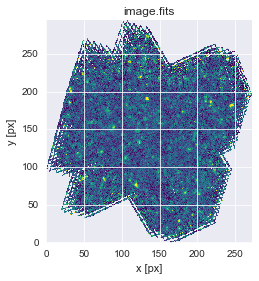
fig, ax = P.subplots()
ax.imshow(ima, cmap='viridis', origin='lower', interpolation='None', vmin=-2e-2, vmax=5e-2)
ax.set_xlabel("x [px]")
ax.set_ylabel("y [px]")
ax.set_title(filename);
|
|
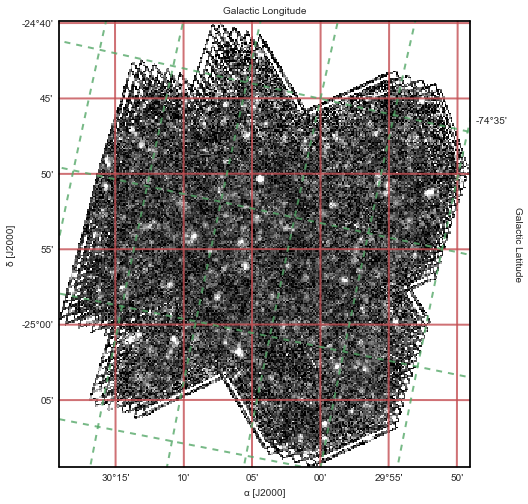
Il est possible d'ajouter d'autres systèmes de coordonnées via WCS.
Requiert WCSaxes, qui n'est pas encore intégré d'office dans astropy.
In [12] :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
25.
26.
27.
28.
29.
30.
31.
32.
import astropy.visualization as VIZ
from astropy.visualization.mpl_normalize import ImageNormalize
fig = P.figure(figsize=(8, 8))
ax = fig.add_subplot(1, 1, 1, projection=wcs)
# 10th and 99th percentiles
vmin, vmax = VIZ.AsymmetricPercentileInterval(10, 99).get_limits(ima)
# Linear normalization
norm = ImageNormalize(vmin=vmin, vmax=vmax, stretch=VIZ.LinearStretch())
ax.imshow(ima, cmap='gray', origin='lower', interpolation='None', norm=norm)
# Coordonnées équatoriales en rouge
ax.coords['ra'].set_axislabel(u'α [J2000]')
ax.coords['dec'].set_axislabel(u'δ [J2000]')
ax.coords['ra'].set_ticks(color='r')
ax.coords['dec'].set_ticks(color='r')
ax.coords.grid(color='r', ls='-', lw=2)
# Coordonnées galactiques en vert
overlay = ax.get_coords_overlay('galactic')
overlay['l'].set_axislabel('Galactic Longitude')
overlay['b'].set_axislabel('Galactic Latitude')
overlay['l'].set_ticks(color='g')
overlay['b'].set_ticks(color='g')
overlay.grid(color='g', ls='--', lw=2)
V-B-2-a-iv. Tables▲
Outre les tables FITS, astropy permet lire et écrire des Tables dans de nombreux formats ASCII usuels en astronomie (LaTeX, HTML, CDS, SExtractor, etc.).
Le fichier ASCII de test est disponible ici : sources.dat.
In [13] :
2.
3.
4.
from astropy.io import ascii
catalog = ascii.read('sources.dat')
catalog.info()
Out [13] :
<Table length=167>
name dtype
------------ -------
ra float64
dec float64
x float64
y float64
raPlusErr float64
decPlusErr float64
raMinusErr float64
decMinusErr float64
xPlusErr float64
yPlusErr float64
xMinusErr float64
yMinusErr float64
flux float64
fluxPlusErr float64
fluxMinusErr float64
background float64
bgPlusErr float64
bgMinusErr float64
quality float64/usr/local/lib/python2.7/dist-packages/astropy/table/column.py:263: FutureWarning: elementwise comparison failed; returning scalar instead, but in the future will perform elementwise comparison return self.data.__eq__(other)
/usr/local/lib/python2.7/dist-packages/astropy/table/info.py:93: UnicodeWarning: Unicode equal comparison failed to convert both arguments to Unicode - interpreting them as being unequal if np.all(info[name] == ''):
In [14] :
2.
3.
4.
5.
catalog.sort('flux') # Ordonne les sources par 'flux' croissant
catalog.reverse() # Ordonne les sources par 'flux' décroissant
#catalog.show_in_notebook(display_length=5)
catalog[:5] # Les 5 sources les plus brillantes du catalogue
Out [14] : <Table length=5>
|
ra float64 |
dec
|
x float64 |
y float64 |
raPlusErr float64 |
|---|---|---|---|---|
|
30.0736543481 |
-24.8389847181 |
133.076596062 |
190.787365523 |
0.000137260656107 |
|
30.0997563127 |
-25.030193106 |
118.886083699 |
76.0608399428 |
0.000200560356177 |
|
30.2726211379 |
-25.0771584874 |
24.9485847511 |
47.803308157 |
0.000495906932571 |
|
29.8763509609 |
-24.7518860739 |
240.583543382 |
242.969136003 |
0.000363715086376 |
|
29.8668948822 |
-24.8539846811 |
245.642862323 |
181.701895847 |
0.000391871197269 |
|
decPlusErr
|
RaMinusErr
|
DecMinusE
|
XplusErr
|
decPlusErr
|
|---|---|---|---|---|
|
0.000124562849667 |
0.000137260656103 |
0.000124562850274 |
0.0747378051241 |
0.000124562849667 |
|
0.00018172429472 |
0.000200560356813 |
0.000181724292183 |
0.109035119289 |
0.00018172429472 |
|
0.000449161858974 |
0.00049590696235 |
0.000449161837629 |
0.269501203678 |
0.000449161858974 |
|
0.000330300804947 |
0.000363715070463 |
0.000330300815023 |
0.198183052816 |
0.000330300804947 |
|
0.000355578581107 |
0.000391871177911 |
0.000355578584919 |
0.213348701017 |
0.000355578581107 |
|
YplusErr
|
XminusErr
|
YminusErr
|
Flux
|
Flux
|
|---|---|---|---|---|
|
0.0747378051241 |
0.0747378051241 |
0.0747378051241 |
157.862010775 |
6.55458899877 |
|
0.109035119289 |
0.109035119289 |
0.109035119289 |
118.448614593 |
7.17503267878 |
|
0.269501203678 |
0.269501203678 |
0.269501203678 |
115.452316575 |
17.2858546025 |
|
0.198183052816 |
0.198183052816 |
0.198183052816 |
95.2024034098 |
10.4819460796 |
|
0.213348701017 |
0.213348701017 |
0.213348701017 |
73.0101287468 |
8.65367562734 |
|
Flux
|
Background
|
BgPlusErr
|
BgMinusErr
|
Quality
|
|---|---|---|---|---|
|
6.55458899877 |
-0.00492962840045 |
0.00280563968109 |
0.00280563968109 |
24.0841967062 |
|
7.17503267878 |
0.00226922676457 |
0.00310958937187 |
0.00310958937187 |
16.5084425251 |
|
17.2858546025 |
0.0108994941326 |
0.00603800958334 |
0.00603800958334 |
6.67900541976 |
|
10.4819460796 |
-0.0013197765479 |
0.00466051306854 |
0.00466051306854 |
9.08251222505 |
|
8.65367562734 |
0.0147626682141 |
0.00330155226713 |
0.00330155226713 |
8.43689223988 |
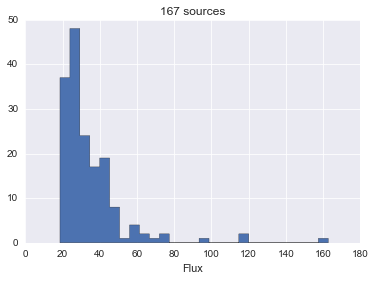
Histogramme des flux, avec choix automatique du nombre de bins :
In [15] :
|
|
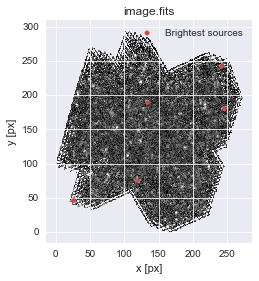
Après conversion des coordonnées RA-Dec de la table en coordonnées, on peut superposer la position des cinq sources les plus brillantes du catalogue à l'image précédente :
In [16] :
2.
3.
4.
5.
6.
7.
fig, ax = P.subplots()
ax.imshow(ima, cmap='gray', origin='lower', interpolation='None', vmin=-2e-2, vmax=5e-2)
ax.set(xlabel="x [px]", ylabel="y [px]", title=filename)
x, y = wcs.wcs_world2pix(catalog['ra'][:5], catalog['dec'][:5], 0)
ax.scatter(x, y, color='r', label="Brightest sources")
ax.legend();
|
|
V-B-2-a-v. Quantités et unités▲
Astropy permet de définir des Quantités dimensionnées et de gérer les conversions d'unités.
In [17] :
2.
3.
4.
from astropy import units as U
from astropy import constants as K
print("Vitesse de la lumière: {} = {}".format(K.c, K.c.to("Mpc/Ga")))
Out [17] :
Vitesse de la lumière: 299792458.0 m / s = 306.601393788 Mpc / Ga
In [18] :
2.
H0 = 70 * U.km / U.s / U.Mpc
print ("H0 = {} = {} = {} = {}".format(H0, H0.to("nm/(a*km)"), H0.to("Mpc/(Ga*Gpc)"), H0.decompose()))
Out [18] :
H0 = 70.0 km / (Mpc s) = 71.5898515538 nm / (a km) = 71.5898515538 Mpc / (Ga Gpc) = 2.26854550263e-18 1 / s
In [19] :
2.
3.
4.
5.
6.
l = 100 * U.micron
print("{} = {}".format(l, l.to(U.GHz, equivalencies=U.spectral())))
s = 1 * U.mJy
print ("{} = {} à {}".format(s, s.to('erg/(cm**2 * s * angstrom)',
equivalencies=U.spectral_density(1 * U.micron)), 1 * U.micron))
Out [19] :
100.0 micron = 2997.92458 GHz
1.0 mJy = 2.99792458e-16 erg / (Angstrom cm2 s) à 1.0 micronV-B-2-a-vi. Calculs cosmologiques▲
Astropy permet des calculs de base de cosmologie.
In [20] :
from astropy.cosmology import Planck15 as cosmo
print(cosmo)Out [20] :
FlatLambdaCDM(name="Planck15", H0=67.7 km / (Mpc s), Om0=0.307, Tcmb0=2.725 K, Neff=3.05, m_nu=[ 0. 0. 0.06] eV, Ob0=0.0486)In [21] :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
25.
26.
27.
28.
29.
30.
31.
32.
33.
34.
35.
36.
37.
38.
39.
40.
41.
42.
43.
44.
45.
46.
47.
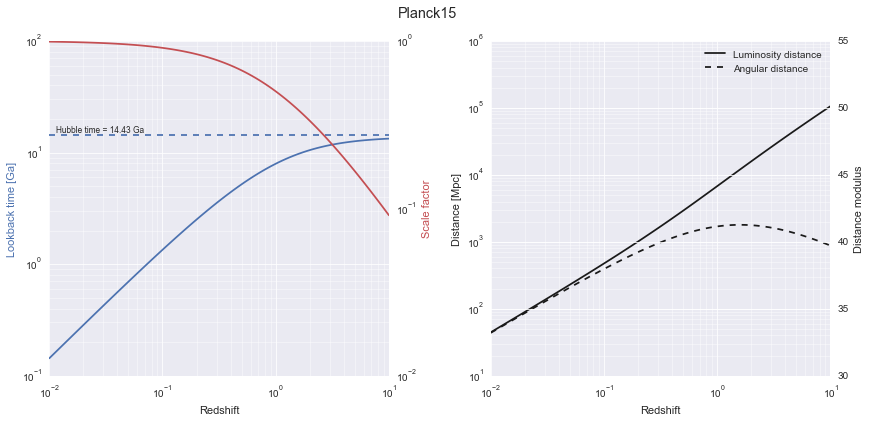
z = N.logspace(-2, 1, 100)
fig = P.figure(figsize=(14, 6))
# Ax1: lookback time
ax1 = fig.add_subplot(1, 2, 1,
xlabel="Redshift", xscale='log')
ax1.plot(z, cosmo.lookback_time(z), 'b-')
ax1.set_ylabel("Lookback time [Ga]", color='b')
ax1.set_yscale('log')
ax1.xaxis.set_minor_locator(P.matplotlib.ticker.LogLocator(subs=range(2,10)))
ax1.yaxis.set_minor_locator(P.matplotlib.ticker.LogLocator(subs=range(2,10)))
ax1.grid(which='minor', color='w', linewidth=0.5)
# En parallèle : facteur d'échelle
ax1b = ax1.twinx()
ax1b.plot(z, cosmo.scale_factor(z), 'r-')
ax1b.set_ylabel("Scale factor", color='r')
ax1b.set_yscale('log')
ax1b.grid(False)
ht = (1/cosmo.H0).to('Ga') # Hubble time
ax1.axhline(ht.value, c='b', ls='--')
ax1.annotate("Hubble time = {:.2f}".format(ht), (z[1], ht.value), (3,3),
textcoords='offset points', size='small');
# Ax2: distances de luminosité et angulaire
ax2 = fig.add_subplot(1, 2, 2,
xlabel="Redshift", xscale='log')
ax2.plot(z, cosmo.luminosity_distance(z), 'k-', label="Luminosity distance")
ax2.plot(z, cosmo.angular_diameter_distance(z), 'k--', label="Angular distance")
ax2.set_ylabel("Distance [Mpc]")
ax2.set_yscale('log')
ax2.legend()
ax2.xaxis.set_minor_locator(P.matplotlib.ticker.LogLocator(subs=range(2,10)))
ax2.yaxis.set_minor_locator(P.matplotlib.ticker.LogLocator(subs=range(2,10)))
ax2.grid(which='minor', color='w', linewidth=0.5)
# En parallèle, module de distance
ax2b = ax2.twinx()
ax2b.plot(z, cosmo.distmod(z), 'k-', visible=False) # Just to get the scale
ax2b.set_ylabel("Distance modulus")
fig.subplots_adjust(wspace=0.3)
fig.suptitle(cosmo.name, fontsize='x-large');
Voir également :
N'oubliez pas de citer [Astropy13] ou de mentionner l'utilisation d'astropy dans vos publications et présentations.
V-C. Autres bibliothèques scientifiques▲
Python est maintenant très largement utilisé par la communauté scientifique, et dispose d'innombrables bibliothèques dédiées aux différents domaines de la physique, chimie, etc. :
- Astronomie : Kapteyn, AstroML, HyperSpy ;
- Mécanique quantique : QuTiP ;
- Électromagnétisme : EMpy ;
- PDE solver : FiPy, SfePy ;
- Analyse statistique bayésienne : PyStan ;
- Markov Chain Monte-Carlo : emcee, PyMC3 ;
- Machine Learning : mlpy, milk, MDP, et autres modules d'intelligence artificielle ;
- Calcul symbolique : sympy (voir également ce tutoriel sympy) et sage ;
- PyROOT ;
- High Performance Computing in Python ;
- Etc.